È l'autunno del 2013 quando, a seguito di innumerevoli segnalazioni di strani disseccamenti sulle chiome degli ulivi, viene identificata la presenza di Xylella fastidiosa da parte di ricercatori del CNR di Bari e dell'Università degli Studi di Bari, e localizzato il primo focolaio di infezione nelle campagne del comune di Gallipoli.
Da allora la presenza sul territorio di questo batterio da quarantena ha determinato l'avvio di una serie di attività di contrasto, con interventi volti a controllare la presenza degli insetti vettore e a ridurre l'inoculo batterico (con l'abbattimento delle piante) per rallentare la diffusione del patogeno Xylella, in attesa di una cura efficace contro il batterio o della selezione di piante resistenti da parte della ricerca scientifica. Questo perché le dimensioni del territorio infetto, la modalità di trasmissione attraverso insetti, quali per esempio la sputacchina (Philaenus spumarius, specie ampiamente polifaga e ubiquitaria), e la vasta gamma di specie vegetali ospiti (molte asintomatiche), non rendono possibile eradicare il patogeno.
Nell'Università del Salento vari gruppi di ricerca sono impegnati nello studio della malattia. Obiettivo principale è comprendere i meccanismi che regolano la risposta delle piante di olivo all'infezione da Xylella fastidiosa; in altre parole spiegare perché alcune varietà mostrano sintomi minori rispetto ad altre. Allo scopo sono state confrontate piante resistenti (cultivar Leccino) con piante sensibili (le cultivar tipiche del Salento, Cellina di Nardò e Ogliarola) attraverso analisi metabolomiche, microscopiche e metagenomiche.
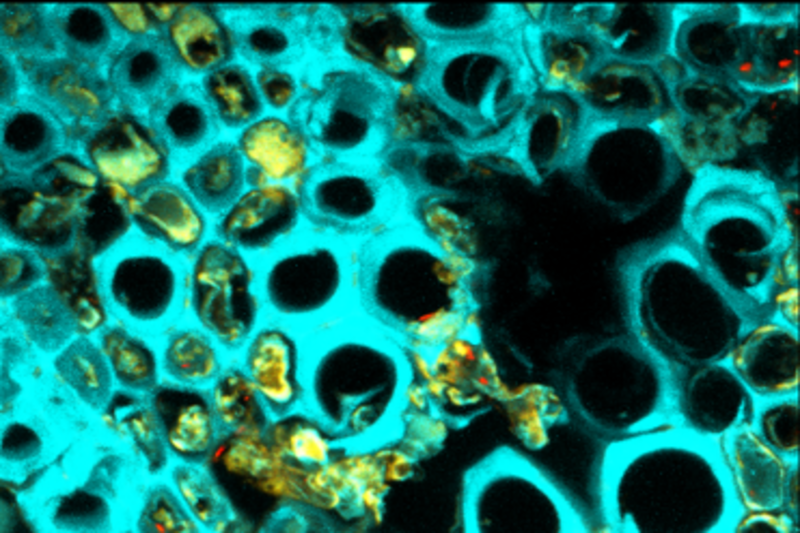
Attraverso la metabolomica (fig. 1) sono stati definiti i profili metabolici, ovvero le sostanze che la pianta produce per contrastare l'infezione, sia agendo direttamente sul patogeno (sostanze antimicrobiche) sia producendo molecole precursori di strutture ¿barriera¿ a cui la pianta ricorre per bloccare, o ridurre, l'ingresso del patogeno o per confinarlo in specifici comparti. La microscopia (fig. 2) ha consentito di studiare l'interazione pianta-patogeno mediante l'osservazione, attraverso una tecnica denominata FISH (Ibridazione Fluorescente In Situ) basata su sonde fluorescenti in grado di legarsi in modo specifico al DNA di Xylella, della distribuzione del batterio nei vasi xilematici (fig. 3) così da evidenziare le differenze nelle modalità di colonizzazione in piante resistenti e suscettibili. Inoltre con tecniche microscopiche sono state studiate le differenze nell'anatomia e nella composizione dei vasi xilematici. Infine, la metagenomica (fig. 4) ha permesso di individuare quali microrganismi siano parte della biodiversità microbica in olivi resistenti e suscettibili, e quali specifici generi o specie microbiche possano svolgere un ruolo attivo nel contrasto alla infezione da Xylella.
Comprendere i fattori alla base della resistenza apre la strada, nel breve periodo, alla selezione mirata di cultivar resistenti, e nel medio-lungo periodo ad attività di miglioramento genetico attraverso incroci non solo per realizzare reimpianti ma anche per ripristinare la biodiversità olivicola sul territorio.
In questo ambito sono state selezionate nel Salento alcune piante di olivo, di cultivar diverse da quelle più diffuse e note, in quanto caratterizzate da una chioma asintomatica nonostante siano circondate da piante evidentemente infette e sintomatiche; queste piante rappresentano sia un ottimo modello di studio che cultivar molto promettenti per la resistenza a Xylella e, quindi, potenzialmente adatte per futuri reimpianti.
Infine, accanto agli studi sui caratteri di resistenza, presso UniSalento procedono parallele altre linee di ricerca, quali lo studio dell'impatto sui servizi eco-sistemici e sul clima dei danni causati da Xylella mediante GIS (Sistema Informativo Geografico) e gli studi indirizzati a realizzare sistemi diagnostici miniaturizzati da utilizzare in campo per una rapida rilevazione del patogeno.